-
V100-gpu_2l和gpu_4l分区计算会爆炸
https://github.com/google-deepmind/alphafold3/issues/59
2024-11-25 admin 0
-
alphafold3--cif格式汇总
CIF文件的基本结构和内容CIF文件由以下几个部分组成:文件头部:包含文件的来源、版本、日期、作者、引用等元数据,以及一些全局的设置和定义。数据块:包含了一个或多个晶体结构的具体数据,每个数据块以“data_”开头,后面跟着一个唯一的标识符。数据块中的数据可以分为标签和值,标签是以“_”开头的预定义或自定义的关键字,值是与标签对应的具体数据,可以是数字、字符串、表达式等。数据块中还可以包含
2024-11-22 admin 0
-
第一个结构1tce -alphafold3蛋白质和小肽相互作用json模板
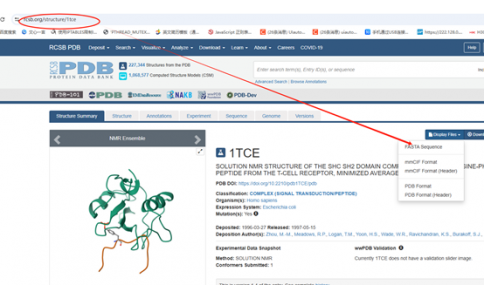
第一个结构-蛋白质和小肽1TCE:一、json结构的准备1、从pdb获取fasta结构2、从实验(测序)获取fasta文件3、从ncbi获取fasta文件4、生成json文件将上述获取的fasta文件,复制到下面文件序列部分(AF3) [chenfj@s61b04n01 testaf3]$ cat 1tce.json{ "name": "1tce", &
2024-11-25 admin 0
-
alphafold3蛋白和小分子相互作用(一)8ssv json模板,小分子结构库里有的(2024年11月结构)
使用https://www.rcsb.org/ligand/H71 的smiles格式计算1、获取小分子信息2、打开网页,3、打开https://www.rcsb.org/fasta/entry/8SSV/display fasta文件4、汇总信息、生成json文件,其中ZN和GOL等基础分子(离子),为默认的ccd格式,是有的,不需要提供smiles格式8ssv.json:{ "
2024-11-22 admin 0
-
alphafold3蛋白和小分子相互作用(二)1w83 json模板 小分子转换为smiles格式
1、截取1w83的小分子存为pdb文件使用 obabel 转化obabel -ipdb 1w83small.pdb -osmi -O 1w83small.smi 如果是mol2格式或者sdf格式obabel -imol2 1w83small.mol2 -osmi -O 1w83small.smiobabel -isdf 1w83small.sdf -osmi -O 1w8
2024-11-22 admin 0
-
alphafold3蛋白和小分子相互作用(三)8R2K json模板 只有cif格式的结构(2024年11月的结构)
1、查看获取小分子信息直接打开 https://www.rcsb.org/ligand/XPI 下载sdf格式wget https://files.rcsb.org/ligands/download/XPI_ideal.sdfobabel -isdf XPI_ideal.sdf -osmi -O 8r2k.smi[chenfj@login26 testnew01]$ cat 8r2k.
2024-11-22 admin 0
-
alphafold3蛋白和小分子相互作用(四)8IT4 蛋白质和小分子共价json模板
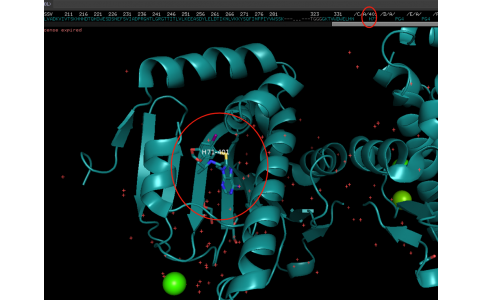
8IT41、获取小分子SKU的cif文件https://www.rcsb.org/ligand/SKU如何查看ccdCodes:下图显示是SKU,连接原子数是29。连接的残基是A链的92位置氨基酸下载 https://files.rcsb.org/ligands/download/SKU.cif2、预先准备好json记住:a、bond必须使用cif不能用smiles,而且得自己合进json文件b、
2024-11-25 admin 0
-
alphafold3蛋白质和RNA相互作用(一)6JVX 单链 json模板
从https://www.rcsb.org/fasta/entry/6JVX /display获取fasta文件,生成以下json,提交任务,其他参考蛋白质和小分子的步骤,{ "name": "6JVX", "modelSeeds": [1, 22, 333, 4444, 66666], "sequences&
2024-11-22 admin 0
-
alphafold3蛋白质和DNA相互作用(一)8ilw 双链 json模板
从https://www.rcsb.org/fasta/entry/6JVX /display获取fasta文件,生成以下json,提交任务,其他参考,蛋白质和小分子的步骤(AF3) [chenfj@s61b04n01 testaf3]$ cat 8ilw.json { "name": "8ILW", "modelSeeds": [1
2024-11-22 admin 0
-
rocky8安装alphafold3--本地安装和部署--no dock和非singularity,非虚拟化版本(原创,转载请注明出处)
1、升级显卡驱动为https://developer.nvidia.com/cuda-downloadshttps://developer.download.nvidia.cn/compute/cudnn/redist/cudnn/linux-x86_64/NVIDIA-SMI 565.57.01 Driver Version: 565.57.01 CUDA V
2024-11-25 admin 0