二、在北极星集群上提交RoseTTAFold-All-Atom------fasta格式
在北极星集群上提交RoseTTAFold-All-Atom------fasta格式
一、fasta格式,必须按照标准来定义:
1、蛋白的fasta
>2V5P_1|Chains A, B|CATION-INDEPENDENT MANNOSE-6-PHOSPHATE RECEPTOR|Homo sapiens (9606)
MKSNEHDDCQVTNPSTGHLFDLSSLSGRAGFTAAYSEKGLVYMSICGENENCPPGVGACFGQTRISVGKANKRLRYVDQVLQLVYKDGSPCPSKSGLSYKSVISFVCRPEAGPTNRPMLISLDKQTCTLFFSWHTPLACEQATECSVRNGSSIVDLSPLIHRTGGYEAYDESEDDASDTNPDFYINICQPLNPMHAVPCPAGAAVCKVPIDGPPIDIGRVAGPPILNPIANEIYLNFESSTPCLADKHFNYTSLIAFHCKRGVSMGTPKLLRTSECDFVFEWETPVVCPDEVRMDGCTLTDEQLLYSFNLSSLSTSTFKVTRDSRTYSVGVCTFAVGPEQGGCKDGGVCLLSGTKGASFGRLQSMKLDYRHQDEAVVLSYVNGDRCPPETDDGVPCVFPFIFNGKSYEECIIESRAKLWCSTTADYDRDHEWGFCRHSNSYRTSSIIFKCDEDEDIGRPQVFSEVRGCDVTFEWKTKVVCPPKKLKHHHHHH
>2V5P_2|Chains C, D|INSULIN-LIKE GROWTH FACTOR II|Homo sapiens (9606)
AYRPSETLCGGELVDTLQFVCGDRGFYFSRPASRVSRRSRGIVEECCFRSCDLALLETYCATPAKSE
其中>为序列描述,用|去分割,第一个列是简称,第二个列是链的名称,第三个为具体的描述,低四个为物种
1、 核酸的fasta格式
DNA:
>8OJD_2|Chain B[auth C]|DNA (47-MER)|synthetic construct (32630)
GCCACTACGACACCTTGATCGCCTCGCAGCCGTCCAACCAACTCAAA
>8OJD_3|Chain C[auth D]|DNA (68-MER)|synthetic construct (32630)
ATTTGCTGACCTTTGTTCTGGGGTGAGTTGGTTGGACGGCTGCGAGGCGATCAAGGTGTCGTAGTGGC
>8XBY_1|Chain A[auth I]|DNA (5'-D(P*AP*AP*CP*GP*AP*AP*AP*AP*CP*GP*GP*CP*CP*AP*CP*CP*AP*CP*G)-3')|synthetic construct (32630)
ATCAGAATCCCGGTGCCGAGGCCGCTCAATTGGTCGTAGACAGCTCTAGCACCGCTTAAACGCACGTACGCGCTGTCCCCCGCGTTTTAACCGCCAAGGGGATTACACCCAAGACACCAGGCACGAGACAGAAAAAAACAACGAAAACGGCCACCACG
RNA:有U!
>8S8X_3|Chain C|m7GpppA-RNA (Cap0-RNA)|synthetic construct (32630)
UUA
二、命令格式
RFAA.run [–pymol] [pdb|fasta] [small.pdb|small.mol2]
格式说明
1、 直接跑 pdb结构 : RFAA.run 1tce.pdb
如果想只跑A链 RFAA.run 1tce.pdb:A
2、将所有的蛋白、DNA、RNA合成一个fasta文件 : RFAA.run input.fasta
!!!!这里多个蛋白或核酸链,fasta文件必须合一个文件,会被自动切割几个文件
3、 跑小分子mol2格式 : RFAA.run 1tce.pdb XG4.mol2
RFAA.run input.fasta XG4.mol2
4、 跑小分子pdb格式 : RFAA.run 1tce.pdb XG4.pdb
RFAA.run input.fasta XG4.pdb
5、 跑小分子sdf格式(OE生成的): RFAA.run 1tce.pdb XG4.sdf
openeye(含smiles格式,推荐4,5) RFAA.run input.fasta XG4.sdf
6、 生成pymol的pse文件,和): RFAA.run -pymol1tce 1tce.pdb XG4.sdf
RFAA.run -pymol 1tce 1tce.pdb
7、共价,预生成格式: RFAA.run 1tce.pdb XG4.pdbyaml
共价键的yaml文件生成请参考 https://github.com/baker-laboratory/RoseTTAFold-All-Atom
修改后提交同上
-h, --help 显示帮助信息
-v, --version 显示版本信息
三、提交案例-fasta
案例路径:/gpfs3/database/RFAA/alian
1、下载fasta并保存8qhh_fasta.fasta (建议不要直接使用8qhh.fasta)
https://www.rcsb.org/fasta/entry/8QHH/display
[chenfj@login28 alian]$ cat 8qhh.fasta
>8QHH_1|Chain A|Bet v 1 related allergen|Actinidia deliciosa (3627)
GAITYDMEIPSSISAEKMFKAFVLDGDTIIPKALPHAITGVQTLEGDGGVGTIKLTTFGEGSVHKSVKHRIDGLDKENFTYSYSIIEGGALDVFESISYHIKIVATPDGGCICKNRSIYTPKCDAQVSEEEIKAGKERASGIFKKVEAYLLANPDC
2、 提交任务
1)提交到gpu_4l (800氨基酸以下): pkurun-g4c 1 1 RFAA.run -pymol 8qhh 8qhh_input.fasta
提交脚本
#!/bin/bash
#SBATCH -J RFA002851
#SBATCH -p gpu_4l
#SBATCH -N 1
#SBATCH -o RFA002851_%j.out
#SBATCH -e RFA002851_%j.err
#SBATCH --no-requeue
#SBATCH -A chen_g1
#SBATCH --qos=cheng4c
#SBATCH --gres=gpu:1
#SBATCH --overcommit
#SBATCH --mincpus=7
pkurun RFAA.run -pymol 8qhh 8qhh_input.fasta
2)提交到gpu_2l (800氨基酸以下): pkurun-g2c 1 1 RFAA.run -pymol 8qhh 8qhh_input.fasta
3)提交到gpu_a800(1300氨基酸以下):pkurun-a800 1 1 RFAA.run -pymol 8qhh 8qhh_input.fasta
3)提交到gpu_ah00(1300氨基酸以下):pkurun-h800 1 1 RFAA.run -pymol 8qhh 8qhh_input.fasta
四、结果
1、对log文件分析
(pymol3) [chen@login28 alian]$ cat 8qhh_input.log
#gen pymol by chenfj
pae_inter: nan ##为相互作用的打分13分以下为佳
mean_plddt: 0.856784999370575
mean_pae: 3.524158239364624
pae_prot: 3.524158239364624
#gen pymol by chenfj
8qhh: (2.541062831878662, 155, 2, 2.5724849700927734, 156, 809.0, 156) ##红色为RMSD
runned min 4.5m ## 跑的时间
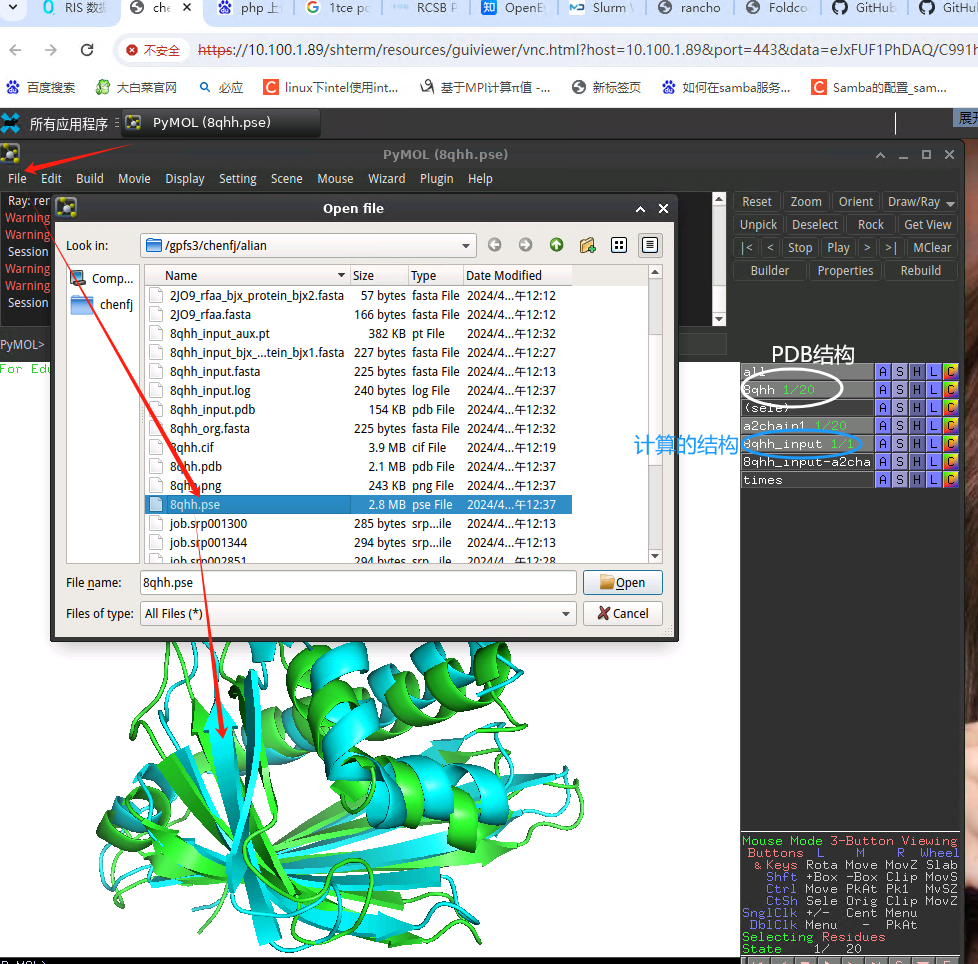
2、打开pse文件
使用北极星图形界面(web)直接打开来看,如下