一、在北极星集群运行alphafold2和结构展示
在北极星集群运行alphafold2:
一、创建并进入lustre3文件夹(lustre1/2/4 gpfs1/2/3,运行比较慢)
mkdir /lustre3/groupname/yourname
二、单GPU执行:
四卡GPU:alphafold-g4c your.fasta
双卡GPU:alphafold-g2c your.fasta
--------------------------------------------------------------------
例如: alphafold-g4c 1tce.fasta
daemo:
1、1TCE daemo:运行命令: alphafold-g4c daemo_1tce
daemo pdb文件 在example/1tce.pdb ![]() 1tce.pdb(点击下载 ,fasta文件 https://www.rcsb.org/fasta/entry/1TCE/display )
1tce.pdb(点击下载 ,fasta文件 https://www.rcsb.org/fasta/entry/1TCE/display )
2、运行自带的daemo: alphafold-g4c daemo
one GPU maybe better!! lustre3 maybe better!!!!!!!!!!!
三、更多 GPU 使用(不建议使用!!!大序列>1200以上未必能快,详细见后面表格):
双卡:alpha_more_gpu-g4c 2 1tce.fasta
三卡: alpha_more_gpu-g4c 3 1tce.fasta
四卡: alpha_more_gpu-g4c 4 1tce.fasta
双卡服务器双卡: alpha_more_gpu-g2c 2 1tce.fasta
四、生成的文件:
第一次运行,比如alphafold-g4c daemo_1tce
1、会拷贝生成alphafold_yourname_time的文件夹,如alphafold_chen_064041
2、生成job.srp+时间的文件,多个结构预测建议修改后直接提交(sbatch job.srp+时间)这样可能会快很多很多,第一次生成文件后删掉提交任务就可以直接改用自己修改后的提交脚本
[chenfj@login12 test2]$ cat job.srp074306
#!/bin/bash
#SBATCH -J alp074306
#SBATCH -p gpu_4l
#SBATCH -N 1
#SBATCH -o alp074306_%j.out
#SBATCH -e alp074306_%j.err
#SBATCH --no-requeue
#SBATCH -A chen_g1
#SBATCH --qos=cheng4c
#SBATCH --gres=gpu:1
#SBATCH --overcommit
#SBATCH --mincpus=7
hosts=`scontrol show hostname $SLURM_JOB_NODELIST` ;echo $hosts
echo CUDA_VISIBLE_DEVICES=$CUDA_VISIBLE_DEVICES
data=/lustre3/alphafold/alphafold-main/scripts
#data=/lustre3/alphafold/alphafold-main/scripts
#data=/lustre2/alphafold-main/scripts
#data=/gpfs1/files_share/alphafold/alphafold-main/scripts
source /appsnew/source/Miniconda3-py38_4.10.3-alphafold.sh
cd /lustre3/chen/alphafold/test/test2/alphafold_chen_074305
time bash run_alphafold.sh -d $data -o /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/dummy_1tce.fasta/ -m model_1 -f /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/example/1tce.fasta -t 2021-07-30 -g -a $CUDA_VISIBLE_DEVICES
.........
##############
注释一:其中data为所下载的各种文件,在哪个文件系统运行就会对应哪个文件系统,比如这次运行在lustre3上就是/lustre3/alphafold/alphafold-main/scripts
注释二:run_alphafold.sh可以更改的:
见链接:https://github.com/kalininalab/alphafold_non_docker
注释三:使用GPU比CPU快1倍,1tce的案例单卡GPU运行时间为20分钟左右(2-4卡也是20分钟),CPU运行时间是37分钟左右
注释四:输出在err,里面有总速度和每步速度
注释五:多个结构预测的时候,可以在 job.srp074306的........处加入所需要的结构提交比如,这样第二次以后的计算会快很多很多:
time bash run_alphafold.sh -d $data -o /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/dummy_1shc.fasta/ -m model_1 -f /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/example/1shc.fasta -t 2021-07-30 -g -a $CUDA_VISIBLE_DEVICES
.........
注意改红色的字体,第二次计算的时候会快很多:
改后的文件为job.srp074306:
#!/bin/bash
#SBATCH -J alp074306
#SBATCH -p gpu_4l
#SBATCH -N 1
#SBATCH -o alp074306_%j.out
#SBATCH -e alp074306_%j.err
#SBATCH --no-requeue
#SBATCH -A chen_g1
#SBATCH --qos=cheng4c
#SBATCH --gres=gpu:1
#SBATCH --overcommit
#SBATCH --mincpus=7
hosts=`scontrol show hostname $SLURM_JOB_NODELIST` ;echo $hosts
echo CUDA_VISIBLE_DEVICES=$CUDA_VISIBLE_DEVICES
data=/lustre3/alphafold/alphafold-main/scripts
#data=/lustre3/alphafold/alphafold-main/scripts
#data=/lustre2/alphafold-main/scripts
#data=/gpfs1/files_share/alphafold/alphafold-main/scripts
source /appsnew/source/Miniconda3-py38_4.10.3-alphafold.sh
cd /lustre3/chen/alphafold/test/test2/alphafold_chen_074305
time bash run_alphafold.sh -d $data -o /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/dummy_1tce.fasta/ -m model_1 -f /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/example/1tce.fasta -t 2021-07-30 -g -a $CUDA_VISIBLE_DEVICES
time bash run_alphafold.sh -d $data -o /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/dummy_1shc.fasta/ -m model_1 -f /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/example/1shc.fasta -t 2021-07-30 -g -a $CUDA_VISIBLE_DEVICES
########################
然后使用sbatch提交 sbatch job.srp074306
注释六:
Usage: run_alphafold.sh
Required Parameters:
-d
-o
-m
-f
-t
Optional Parameters:
-b
proteins (default: 'False')
-g
-a
-p
3、/lustre3/chen/alphafold/test/test2/alphafold_chen_074305/example下有pdb结构文件1tce.pdb
4、结果文件夹在/lustre3/chen/alphafold/test/test2/alphafold_chen_074305/dummy_1tce.fasta/下
features.pkl
ranked_{0,1,2,3,4}.pdb
ranking_debug.json
relaxed_model_{1,2,3,4,5}.pdb
result_model_{1,2,3,4,5}.pkl
timings.json
unrelaxed_model_{1,2,3,4,5}.pdb
msas/
bfd_uniclust_hits.a3m
mgnify_hits.sto
uniref90_hits.sto
参考:https://github.com/deepmind/alphafold
5、可以使用pymol比较1tce.pdb和 ranked_{0,1,2,3,4}.pdb去align
下载链接:http://221.216.6.54:808/sys_bio_lib/software/pymol/
6、在 /lustre3/groupname/yourname/alphafold_yourname_time/alphafold/data/tools/_ 下
比如 /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/alphafold/data/tools/
hhblits.py
hhsearch.py
hmmbuild.py
hmmsearch.py
jackhmmer.py
kalign.py
utils.py
可以修改这几个文件里面的参数。比如jackhmmer.py:
def __init__(self,
*,
binary_path: str,
database_path: str,
n_cpu: int = 8,
n_iter: int = 1,
e_value: float = 0.0001,
z_value: Optional[int] = None,
get_tblout: bool = False,
filter_f1: float = 0.0005,
filter_f2: float = 0.00005,
filter_f3: float = 0.0000005,
incdom_e: Optional[float] = None,
dom_e: Optional[float] = None,
num_streamed_chunks: Optional[int] = None,
streaming_callback: Optional[Callable[[int], None]] = None):
7、更多GPU,不建议使用!:
两个卡 alpha_more_gpu-g4c 2 1tce.fasta
其他:
or: alpha_more_gpu-g4c 3 1tce.fasta
or: alpha_more_gpu-g4c 4 1tce.fasta
or: alpha_more_gpu-g2c 2 1tce.fasta
8、daemon_1tce和结果展示:
在lustre3上,用1个GPU卡跑是20分钟,用默认CPU跑是37分钟
在lustre3你的文件夹下运行:
alphafold-g4c daemo_1tce
结果见上面描述:
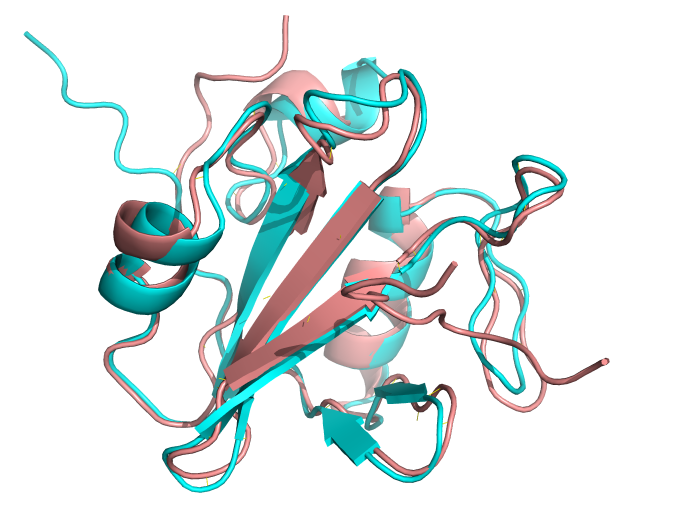
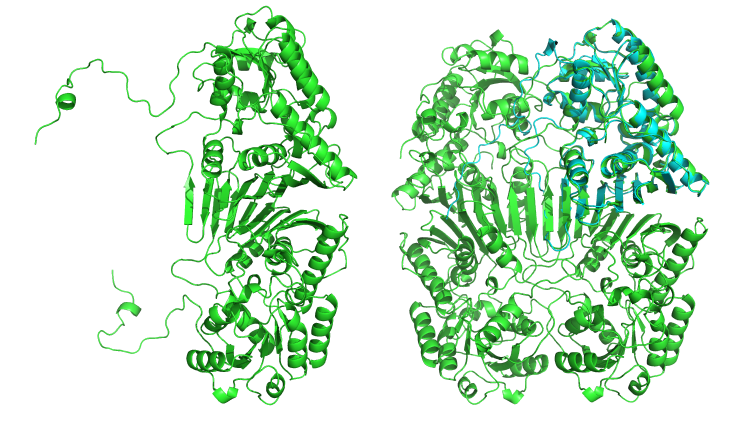
浅棕色是1TCE的晶体结构,青色是预测的结构。在lustre3上,用1个GPU卡跑是20分钟,用默认CPU跑是37分钟
9,不能超过一个蛋白链:
如:
>2JO9_1|Chain A|Itchy E3 ubiquitin protein ligase|Mus musculus (10090) GAMGPLPPGWEKRTDSNGRVYFVNHNTRITQWEDPRS >2JO9_2|Chain B|Latent membrane protein 2|null EEPPPPYED
More than one input sequence found in /lustre3/chen/alphafold/test/test2/alphafold_chen_005/2JOV.fasta
五、多个测试结果,从100个氨基酸到2470个氨基酸的结构:
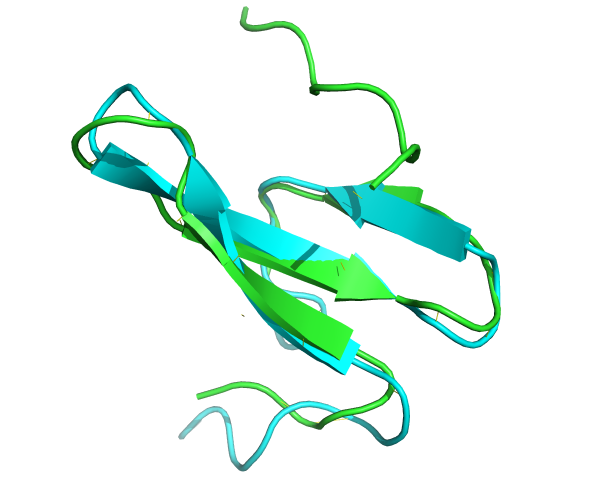
绿色是2JO9的晶体结构,青色是预测的结构 pse文件![]() 2jo9.zip
2jo9.zip
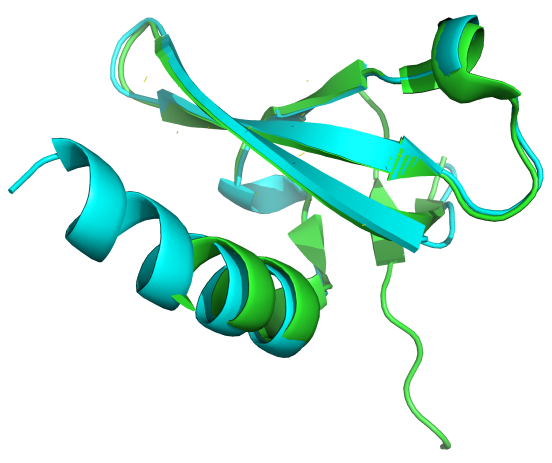
绿色是3FDT的晶体结构,青色是预测的结构 pse文件![]() 3fdt.zip
3fdt.zip
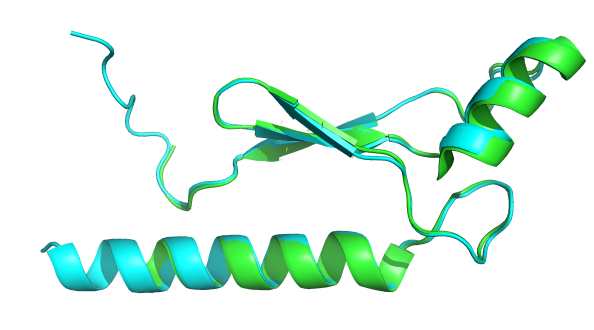
绿色是3DGL的晶体结构,青色是预测的结构 pse文件![]() 3dgl.zip
3dgl.zip
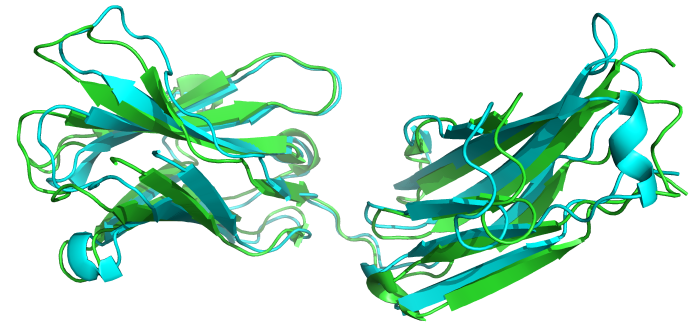
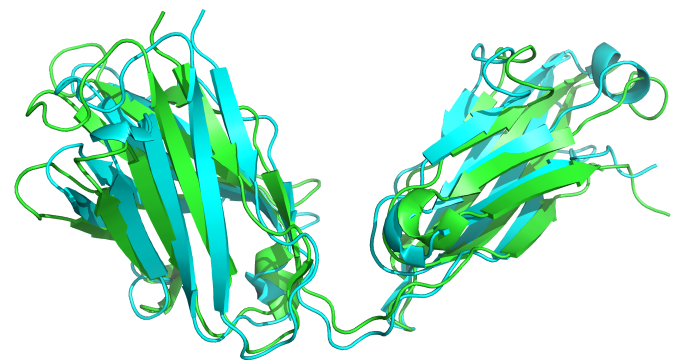
绿色是3B2U的晶体结构,青色是预测的结构 pse文件![]() 3b2u.zip
3b2u.zip
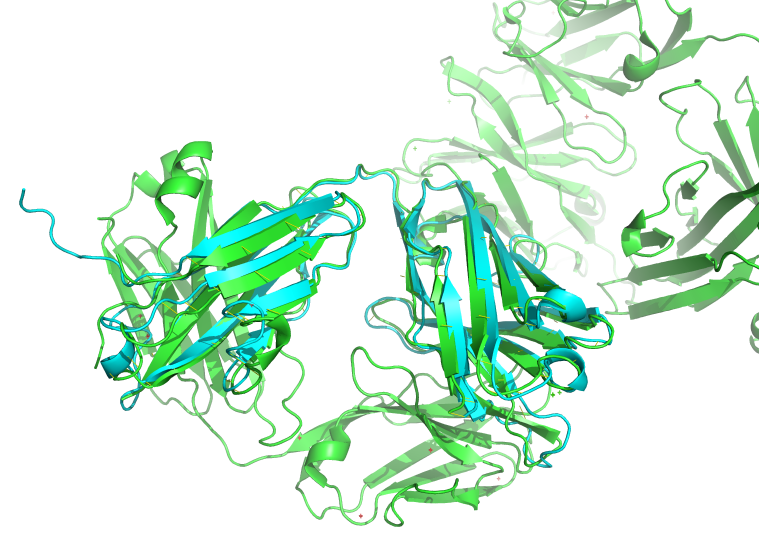
绿色是3B2U的晶体结构,青色是预测的结构 pse文件![]() 3b2u.zip
3b2u.zip

绿色是3AY6的晶体结构,青色是预测的结构 pse文件![]() 3ay6.zip
3ay6.zip
plk1 PTB 3DB6:
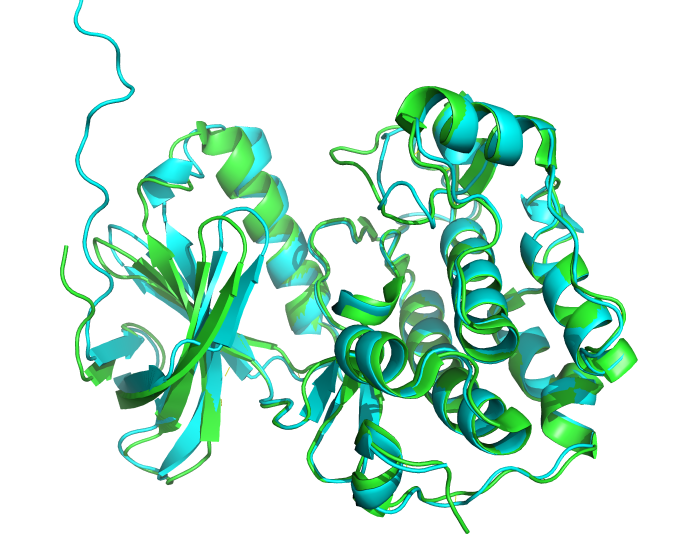
绿色是3DB6的晶体结构,青色是预测的结构 pse文件![]() 3db6.zip
3db6.zip
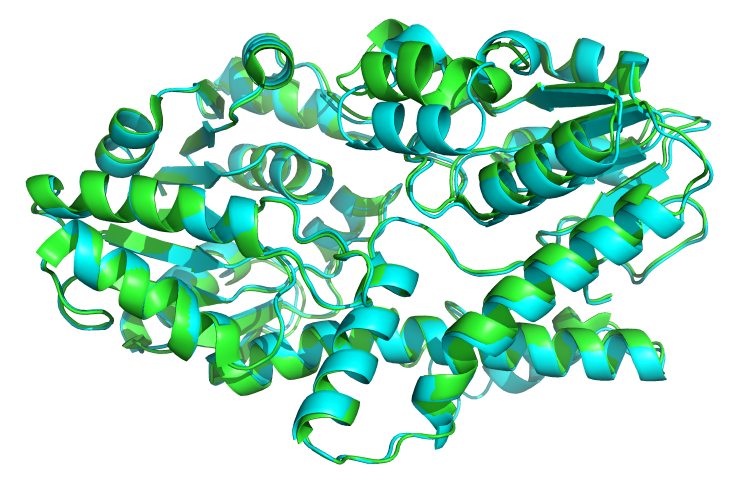
绿色是1OFG的晶体结构,青色是预测的结构 pse文件![]() 1ofg.zip
1ofg.zip
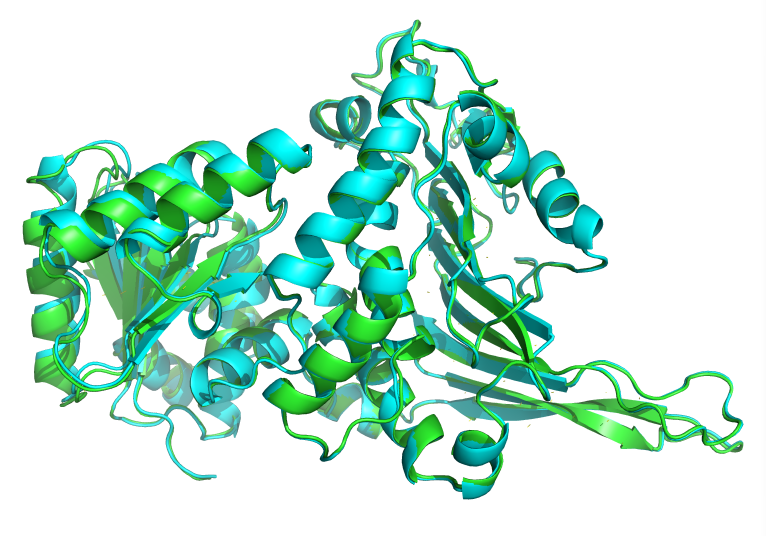
绿色是1MUU的晶体结构,青色是预测的结构 pse文件![]() 1muu.zip
1muu.zip
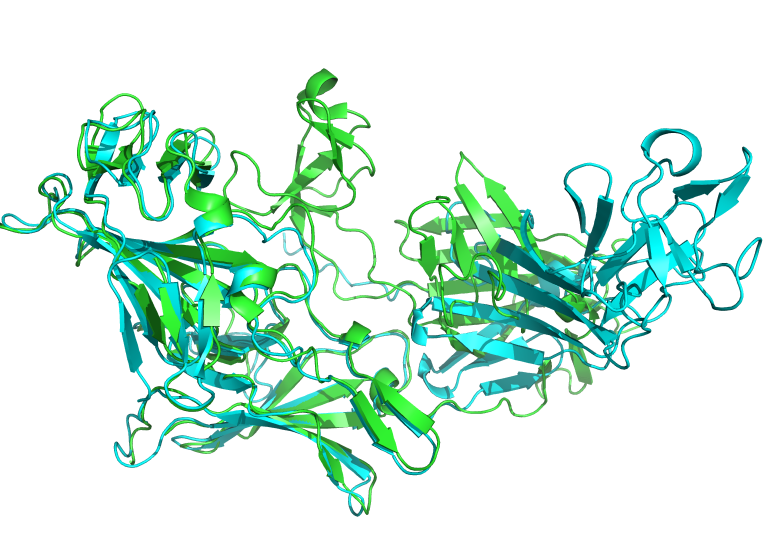
绿色是1E7M的晶体结构,青色是预测的结构 pse文件![]() 1e7m.zip
1e7m.zip
绿色是2V5P的晶体结构,青色是预测的结构 pse结构 ![]() 2v5p.zip
2v5p.zip
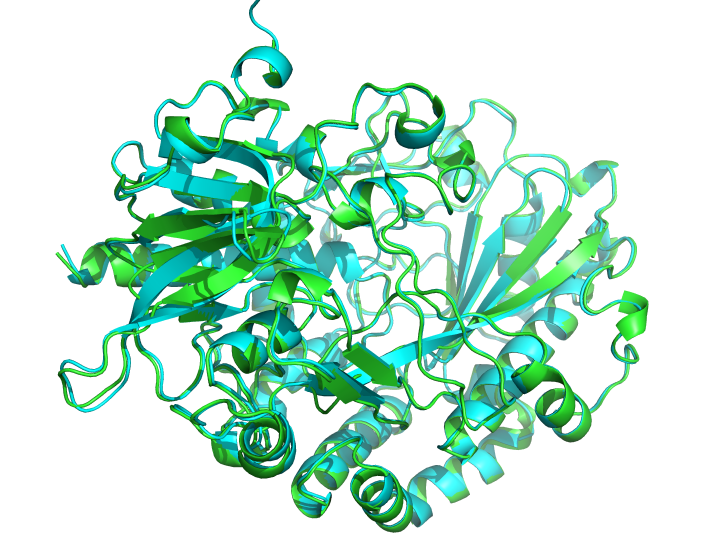
绿色是1CF3的晶体结构,青色是预测的结构 pse结构![]() 1cf3.zip
1cf3.zip
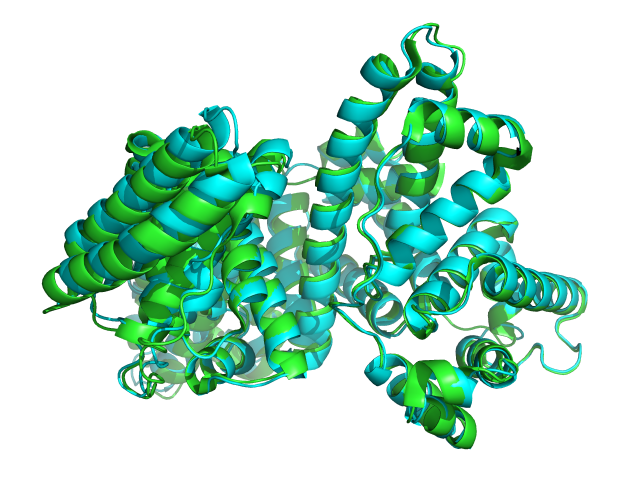
绿色是6HN1的晶体结构,青色是预测的结构 pse结构 ![]() 6hn1.zip
6hn1.zip
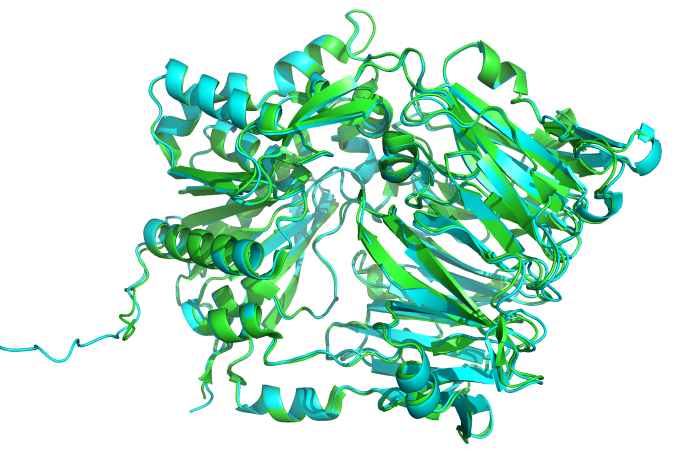
绿色是4A5S的晶体结构,青色是预测的结构 pse结构 ![]() 4a5s.zip
4a5s.zip
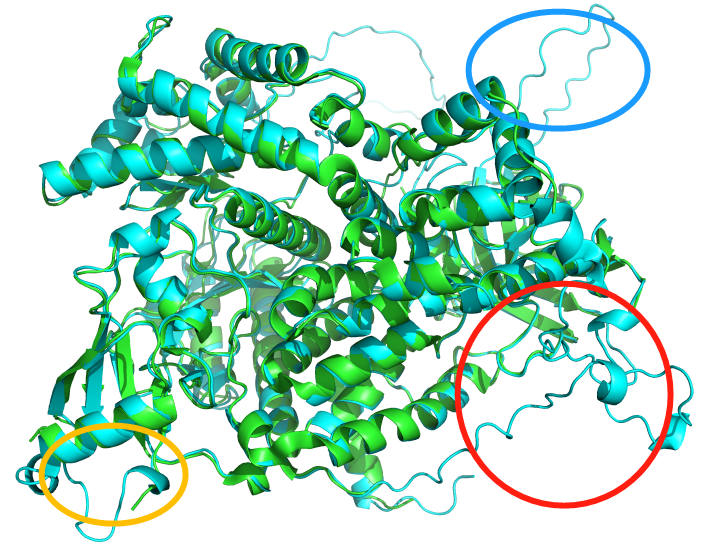
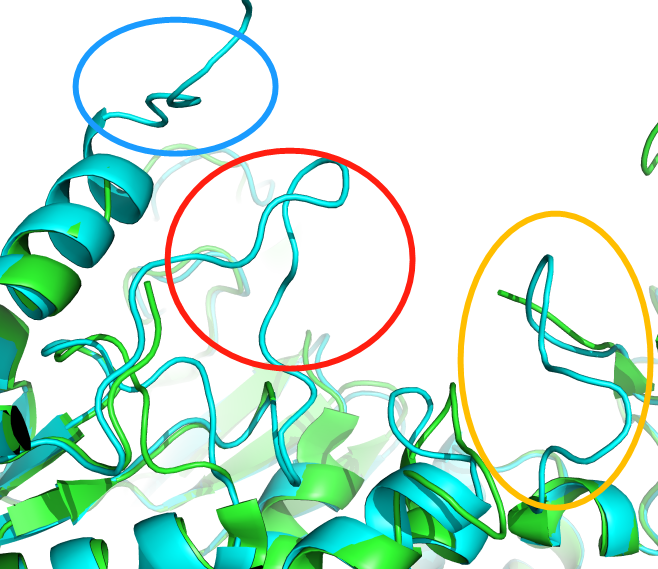
绿色是4ANU的晶体结构,青色是预测的结构,划圈的是补充的结构。 pse结构![]() 4anu.zip
4anu.zip
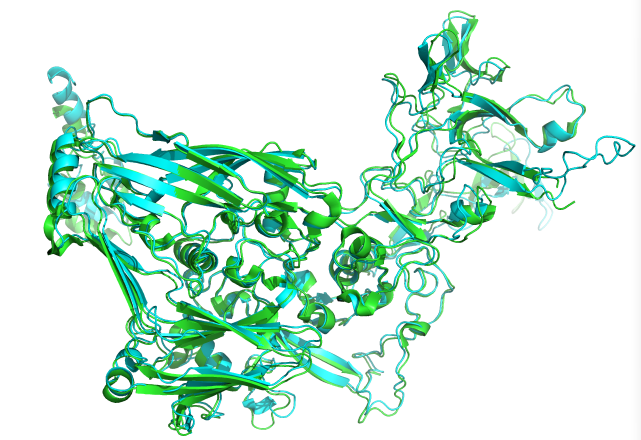
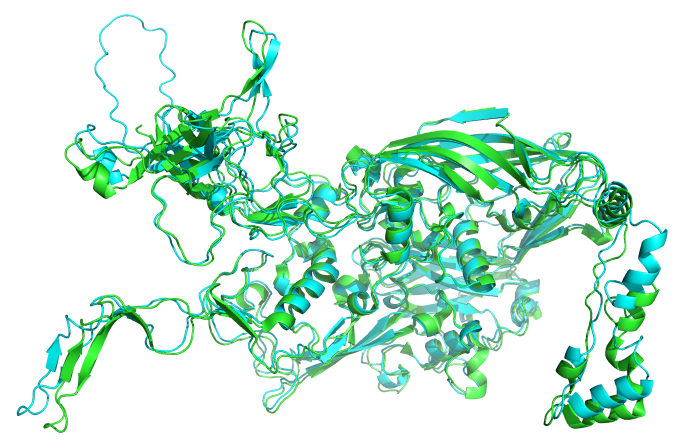
绿色是6EQC的晶体结构,青色是预测的结构 pse结构![]() 6eqc.zip
6eqc.zip
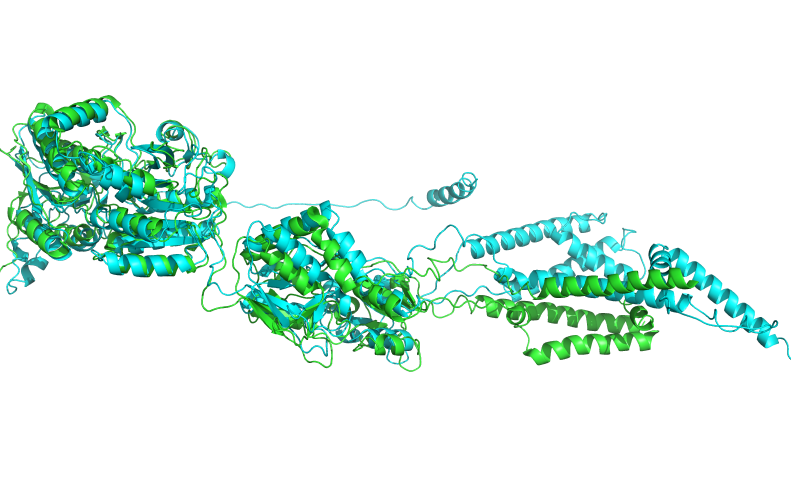
绿色是7LZH的晶体结构,青色是预测的结构pse文件下载 ![]() 71zh.zip
71zh.zip
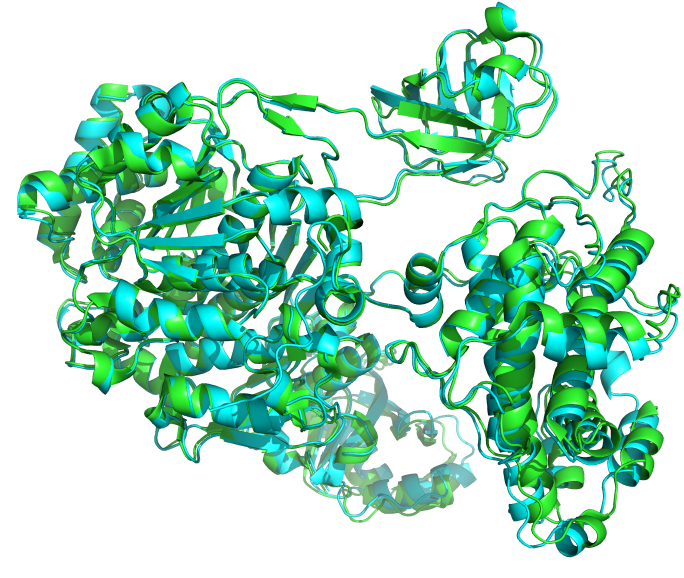
绿色是5L6H的晶体结构,青色是预测的结构,几个连接出比较好,pse文件下载![]() 5l6h.zip
5l6h.zip
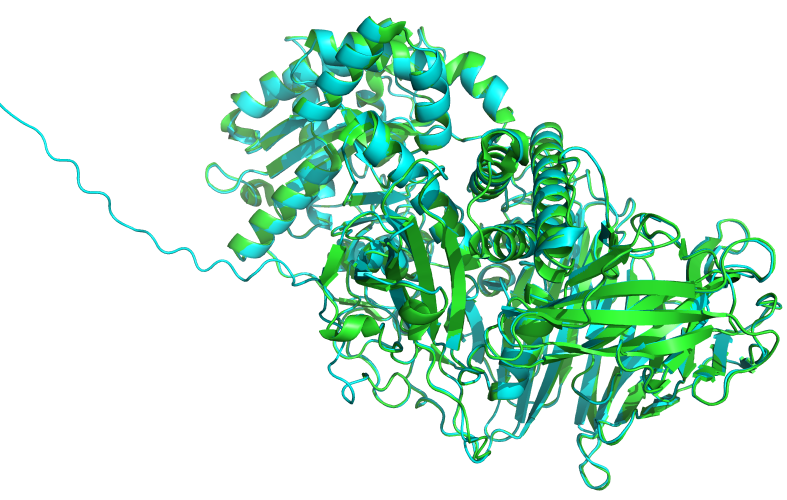
绿色是2F18的晶体结构,青色是预测的结构pse文件下载![]() 2f18.zip
2f18.zip
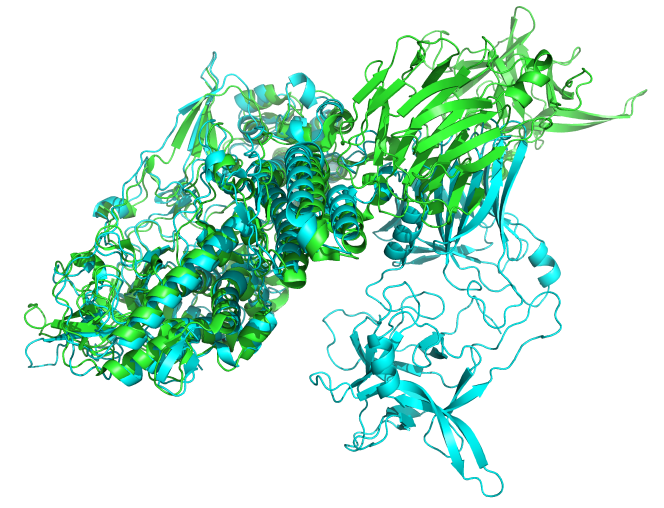
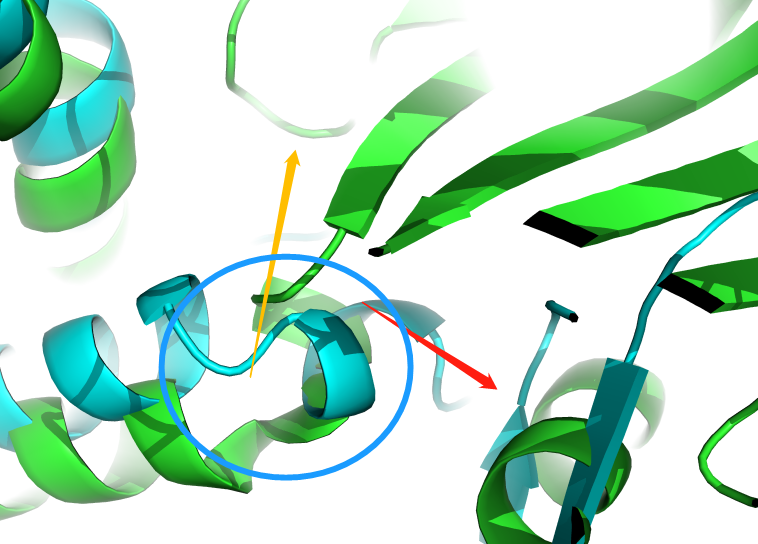
绿色是1G9A的晶体结构,青色是预测的结构,箭头所指示的是不能叠合的原因,α螺旋改变方向。pse文件下载![]() 1g9a.zip
1g9a.zip
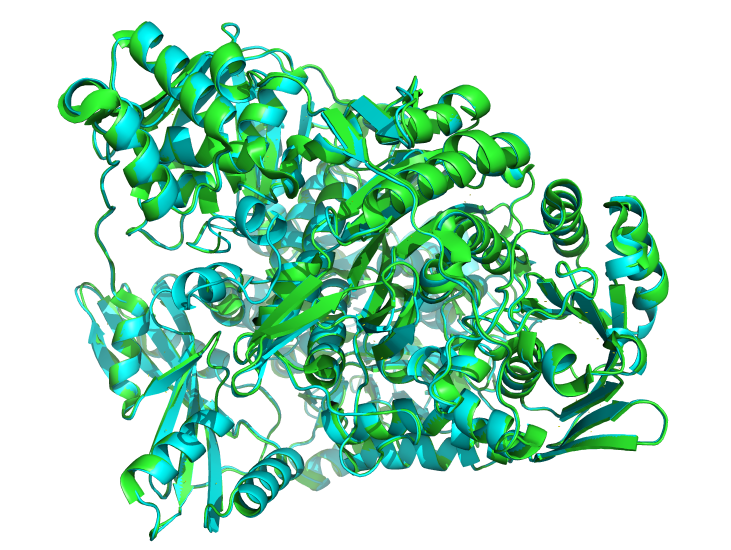
绿色是1T3T的晶体结构,青色是预测的结构pse文件下载![]() 1t3t.zip
1t3t.zip
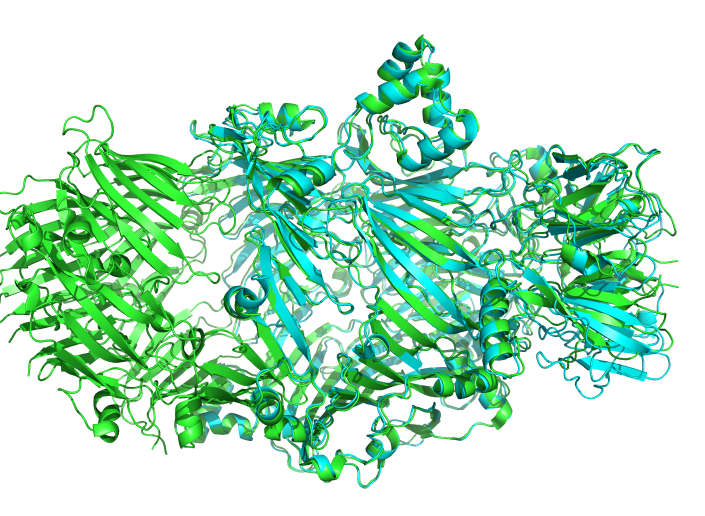
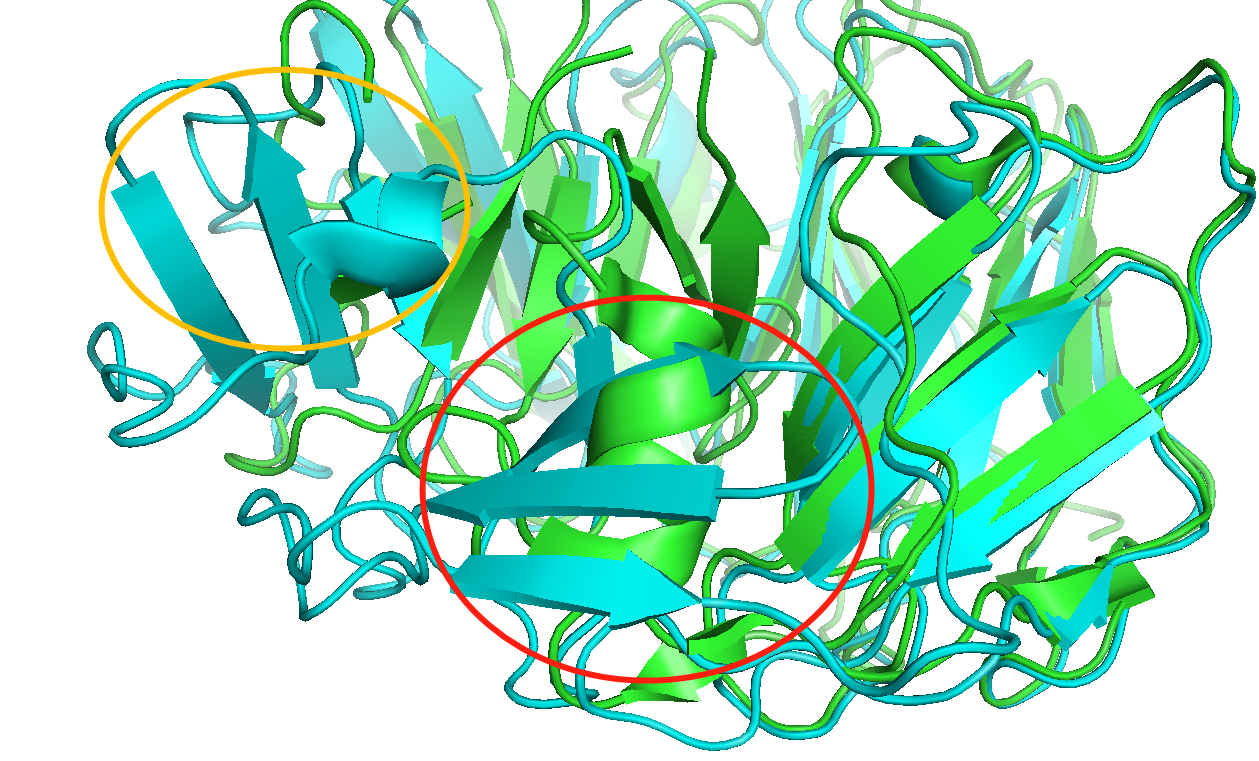
绿色是5KIS的晶体结构,青色是预测的结构pse文件下载 ![]() 5kis.zip
5kis.zip

绿色是6MU1的晶体结构,青色是预测的结构pse文件下载![]() 6mu1.zip
6mu1.zip
| PDB | 2JO9 | 3FDT | 3DGL | 1TCE | 3B2U | 3POV | 3AY6 | 3DB6 | 1OFG | 1MUU | 1E7M | 2V5P | 1CF3 |
| aa number | 33 | 60 | 82 | 108 | 225 | 229 | 270 | 298 | 382 | 437 | 486 | 493 | 584 |
| CPUs(4/7) | 669 | 248 | 206 | 37(Min) | 83 | 76 | 402 | 451 | 193 | 222 | 130 | 245 | 258 |
| 1GPU(Min) | 188 | 694/471 | 195 | 21 | 57 | 61 | 244 | 216 | 118 | 108 | 58 | 37/175 | 121 |
| 2GPU(Min) | 225 | 199 | 190 | 20 | 48 | 61 | 243 | 216 | 98 | 106 | 60 | 29 | 118 |
| 3GPU(Min) | 206 | 198 | 195 | 20 | 45 | 49 | 256 | 216 | 99 | 79 | 59 | 33 | 110 |
| 4GPU(Min) | 208 | 218 | 195 | 21 | 57 | 55 | 243 | 216 | 85 | 85 | 58 | 32 | 98 |
| 大致评价5分 | 4.8 | 5 | 5 | 4.7 | 4.6 | 4.7 | 5 | 4.8 | 4.9 | 4.8 | 5 | 4.0 | 4.9 |
| 原因 | 完全β折叠 | CPU比单GPU快! | 补齐了两端 | β折叠为主骨架 | β折叠比较多 | β折叠比较多 | 天知道为毛跑这么久 | 序列短不一定时间短 | 特别小 比较完美 | β折叠比较多 | α螺旋相对多 | ||
| 可以看见1000氨基酸以内 1卡GPU的计算和多卡还是没有本质区别,序列长短并不是明显决定计算时间的因素,更多的是结构的复杂性和库里的文件多少。α螺旋结构多的比较准确,几乎是β折叠的会差些。GPU加速方面,序列越长相对加速越明显比如6EQC,基本上都有一倍以上的提升,但是速度变化范围比较大,见下划线和后面表格三。红蓝相间为GPU提速比较明显的,4倍以上。 | |||||||||||||
接上表
| PDB | 6HN1 | 4A5S | 4ANU | 6EQC | 7LZH | 5L6H | 3D4Y | 2F18 | 1G9A | 4XWK | 1T3T | 5KIS | 5URK | 6MU1 |
| aa number | 610 | 741 | 833 | 953 | 960 | 1029 | 1046 | 1046 | 1291 | 1295 | 1304 | 1492 | 1739 | 2740 |
| CPUs(4/7) | 276 | 740 | 963 | 379 | 失败 | 失败 | ||||||||
| 1GPU(Min) | 83/167 | 329 | 233/210 | 52 | 165 | 132 | 185 | 180 | 200 | tensor proto larger than 2GB | 89 | 351 | not find 233 match states in seq | 606 |
| 2GPU(Min) | 80 | 325 | 209 | 61 | 155 | 139 | 113 | 191 | 191 | 105 | 349 | 737 | ||
| 3GPU(Min) | 66 | 326 | 207 | 49 | 157 | 100 | 148 | 116 | 178 | 77 | 344 | 353 | ||
| 4GPU(Min) | 67 | 323 | 232 | 60 | 153 | 121 | 86 | 75 | 171 | 148 | 321 | 393 | ||
大致评价 5分 | 4.9 | 4.9 | 5 | 4.9 | 3.7 | 5 | 4.9 | 4.9 | 4.3 | 4.9 | 4.7 | 4.5 | ||
| 原因 | α螺旋多 | β折叠较多,但好 | 补齐了loop等结构 | 不,补齐了loop | 分子大loop多 | 几个连接出比较好 | 结构同2F18 | α螺旋占一半多 | 很大部分不能叠合 | 内存超了! | 大部分α螺旋 | 有部分折叠变 螺旋 | 补了不少结构, | |
| 可以看见1000氨基酸以内 1卡GPU的计算和多卡还是没有本质区别,序列长短并不是明显决定计算时间的因素,更多的是结构的复杂性和库里的文件多少。α螺旋结构多的比较准确,几乎是β折叠的会差些。GPU加速方面,序列越长相对加速越明显比如6EQC,基本上都有一倍以上的提升,但是速度变化范围比较大,见下划线和后面表格。红蓝相间为GPU提速比较明显的。 | ||||||||||||||
PDB 结构 1TCE的结果波动也是很大:表格三
