人类的妊娠效率很低,自然妊娠中只有不到50%的胚胎能够发育至足月。部分流产胚胎主要是源于胚胎的非整倍体染色体异常,在移植胚胎前鉴定并排除非整倍体胚胎是辅助生殖领域面临的巨大挑战。当前临床上多采用对植入前胚胎的滋养外胚层进行样品活检和遗传学检测的方式来分析胚胎细胞的染色体倍性。该方法因涉及侵入性的胚胎细胞活检,操作繁琐,无法避免对胚胎造成一定程度的机械损伤。以检测体外培养胚胎的培养液中游离DNA为代表的无创植入前胚胎遗传学检测,因没有对胚胎本身进行取样操作,能够避免上述问题,但无创检测的一个难点在于对培养液中游离DNA的细胞溯源,这直接关乎无创检测方法的检测准确性。

2021年6月15日,生命科学联合中心、北京大学生物医学前沿创新中心汤富酬、北京大学生物医学前沿创新中心文路团队与生命科学联合中心、北京大学第三医院乔杰、北京大学第三医院黄锦团队合作在The Journal of Clinical Investigation杂志发表题为DNA methylome reveals cellular origin of cell-free DNA in spent medium of human preimplantation embryos的研究论文。该研究对194例体外培养胚胎的培养液(spent embryo culture media,SECM)进行微量DNA甲基化组测序(Post–bisulfite adaptor tagging–based single-cell whole-genome DNA methylation sequencing, scBS-seq),通过和此前该团队合作发表的植入前胚胎的单细胞DNA甲基化组图谱进行系统比较分析,鉴定了培养液中游离DNA(cell free DNA,cfDNA)的准确细胞来源,并在此基础上精准定量了培养液中的来自颗粒细胞的母源DNA污染,进而实现了系统的整合分析(图1)。
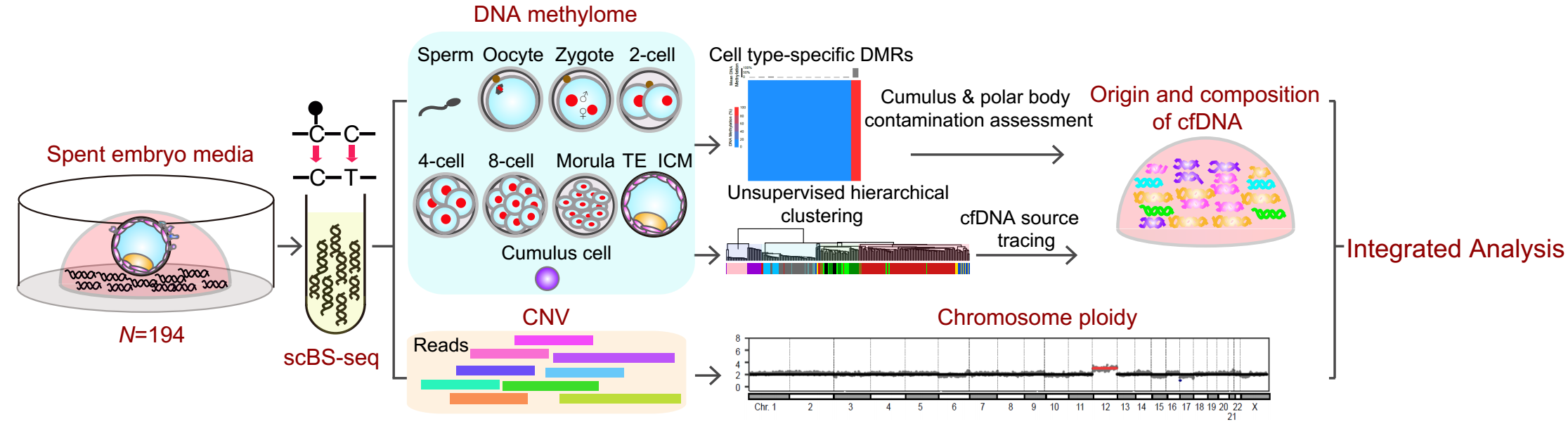
图1 胚胎培养液cfDNA甲基化图谱研究示意图
1.胚胎培养液游离DNA中存在来自母体颗粒细胞的游离DNA污染
该研究将培养液游离DNA的甲基化与植入前胚胎细胞和颗粒细胞基因组DNA的甲基化进行了系统的非监督分析,发现50/191 SECM与颗粒细胞聚为一类,这些SECM呈现较高的全基因组甲基化水平,与颗粒细胞的全基因组甲基化水平接近(图 2A)。
为了进一步量化SECM中颗粒细胞污染来源的DNA比例,研究鉴定了769个颗粒细胞特异高甲基化的差异甲基化区域(C-DMRs),这些差异甲基化区域在颗粒细胞中高度甲基化,而在植入前胚胎细胞基因组DNA中近乎零甲基化(图 2B)。根据C-DMRs的甲基化水平,将不同的SECM样品分为无颗粒细胞污染的样品、颗粒细胞中度污染的样品、颗粒细胞重度污染的样品三组,结果显示随着颗粒细胞污染比例的增加,SECM样品的总体甲基化水平相应增加,表明SECM的总体甲基化水平高很大程度上是由颗粒细胞来源的游离DNA污染引起的(图 2C)。
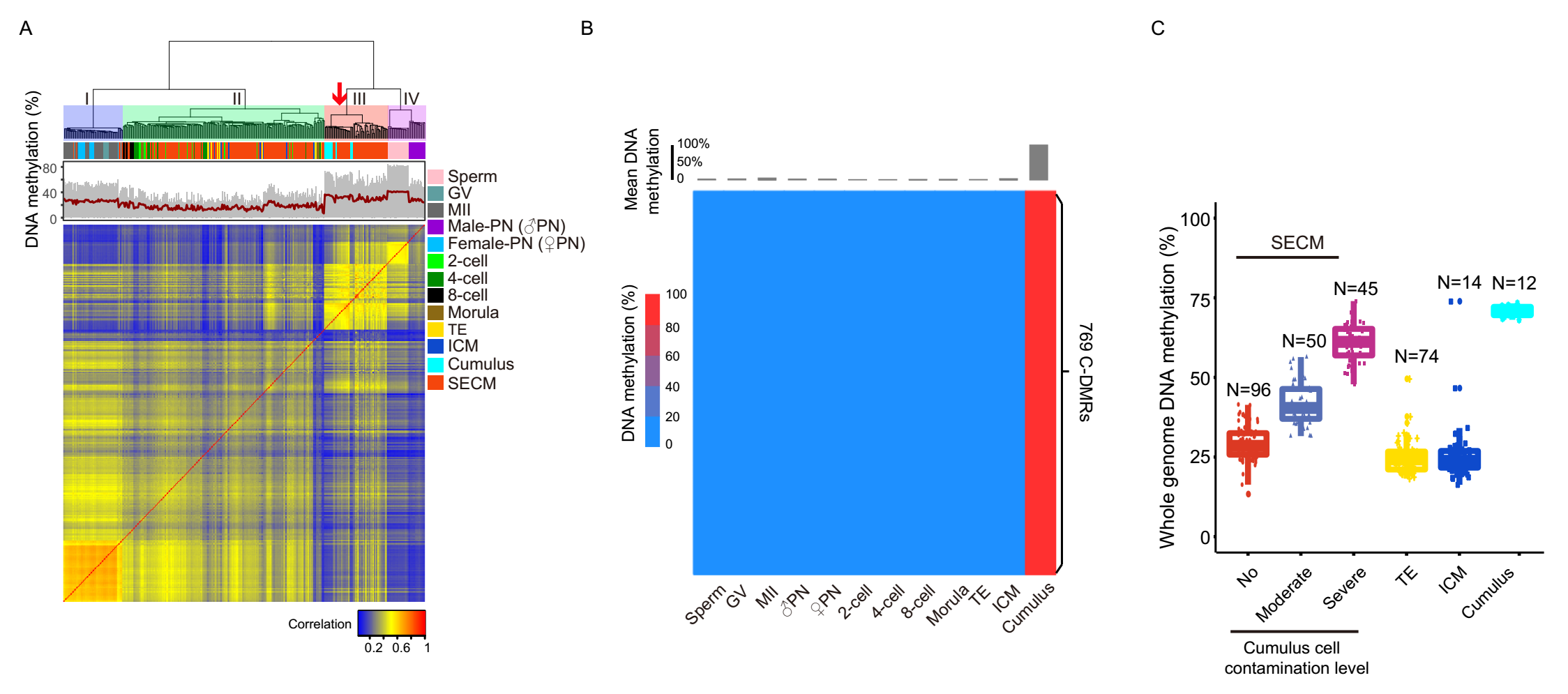
图2 胚胎培养液(SECM)中存在颗粒细胞污染。(A)SECM与植入前胚胎细胞、颗粒细胞非监督聚类分析;(B)769个颗粒细胞特异高甲基化区域(C-DMRs);(C)三类SECM的全基因组甲基化水平。
2.胚胎培养液游离DNA中存在来自胚胎极体细胞的游离DNA污染
为了进一步探究无颗粒细胞污染的SECM中游离DNA的准确来源,该研究继续将培养液游离DNA的甲基化与植入前胚胎细胞的基因组甲基化进行了系统的非监督聚类分析,结果显示3/96 SECM与MII期卵细胞与雌原核的基因组DNA聚为一类(图3A)。由于胚胎在体外培养过程中仍在持续发育,因此卵细胞中雌原核的基因组DNA不会被释放到培养液中,据此推测胚胎培养液游离DNA的来源是排出胚胎外的极体细胞。
为了进一步量化SECM中污染的极体细胞游离DNA的比例,研究鉴定了548个卵/极体细胞特异高甲基化的差异甲基化区域(O-DMRs),这些区域在卵细胞和雌原核中高度甲基化,而在植入前胚胎细胞以及颗粒细胞中低甲基化(图 3B)。据文献报道,非CpG的甲基化水平在卵母细胞中显著高于其他植入前阶段的胚胎细胞。在无颗粒细胞污染的SECM中,CHG和CHH(非CpG)的甲基化水平与O-DMRs甲基化水平呈正相关,表明SECM中有时存在极体细胞污染(图 3C)。
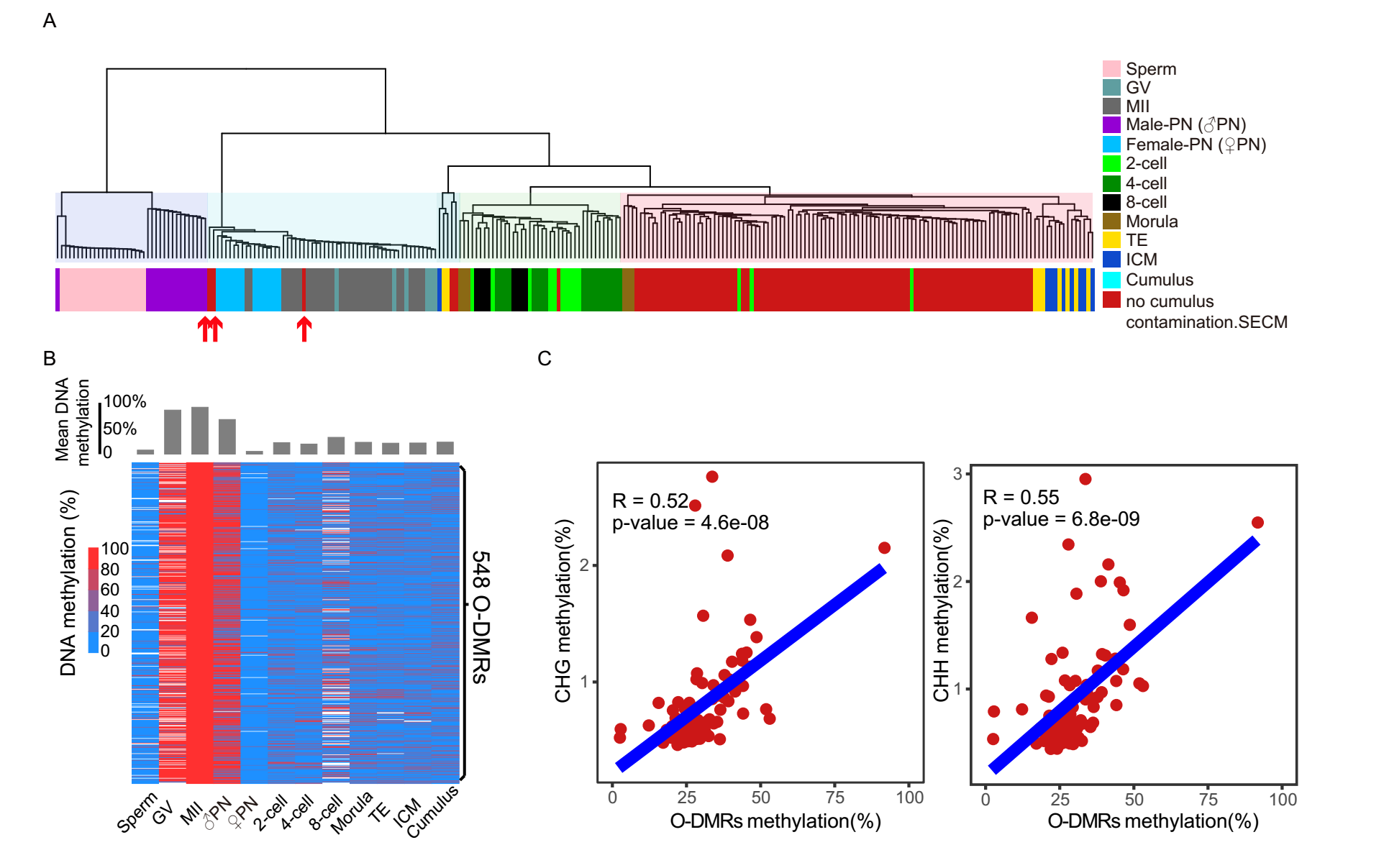
图3 胚胎培养液(SECM)中存在极体细胞污染。(A)无颗粒细胞污染的SECM与植入前胚胎细胞、颗粒细胞非监督聚类分析;(B)548个卵/极体细胞特异高甲基化区域(O-DMRs);(C)无颗粒细胞污染的SECM中非CpG甲基化水平与O-DMRs甲基化水平的关系。
3.胚胎培养液游离DNA中母源DNA污染率的推导
利用769个C-DMRs和548个O-DMRs的甲基化水平数据,该研究团队建立计算方法推导了胚胎培养液游离DNA中颗粒细胞和极体细胞DNA组分的比例。首先,通过计算机生成一系列掺入不同比例的ICM/TE和颗粒细胞、极体细胞的模拟混合数据,再利用该计算方法计算颗粒细胞和极体细胞的百分比,发现计算得到的比例与实际掺入的比例高度吻合,这表明该计算方法的准确性很高(图 4A)。
接着该研究计算了实际培养液游离DNA中各组分的比例,发现1/3的胚胎培养液游离DNA样本中存在颗粒细胞污染,且22%的样本颗粒细胞污染比例超过60%以上。1/3的胚胎培养液游离DNA样本存在极体细胞污染,4%的样本是极体污染超过60%以上。说明通常情况下在胚胎培养液游离DNA中颗粒细胞污染比极体细胞污染更严重。
随后,该研究定义了颗粒细胞和极体细胞污染比例之和为净母源污染,发现只有1/3的胚胎培养液游离DNA样品不存在明显的母源污染,将近1/3的胚胎培养液游离DNA样本存在严重的母源污染(图 4B)。
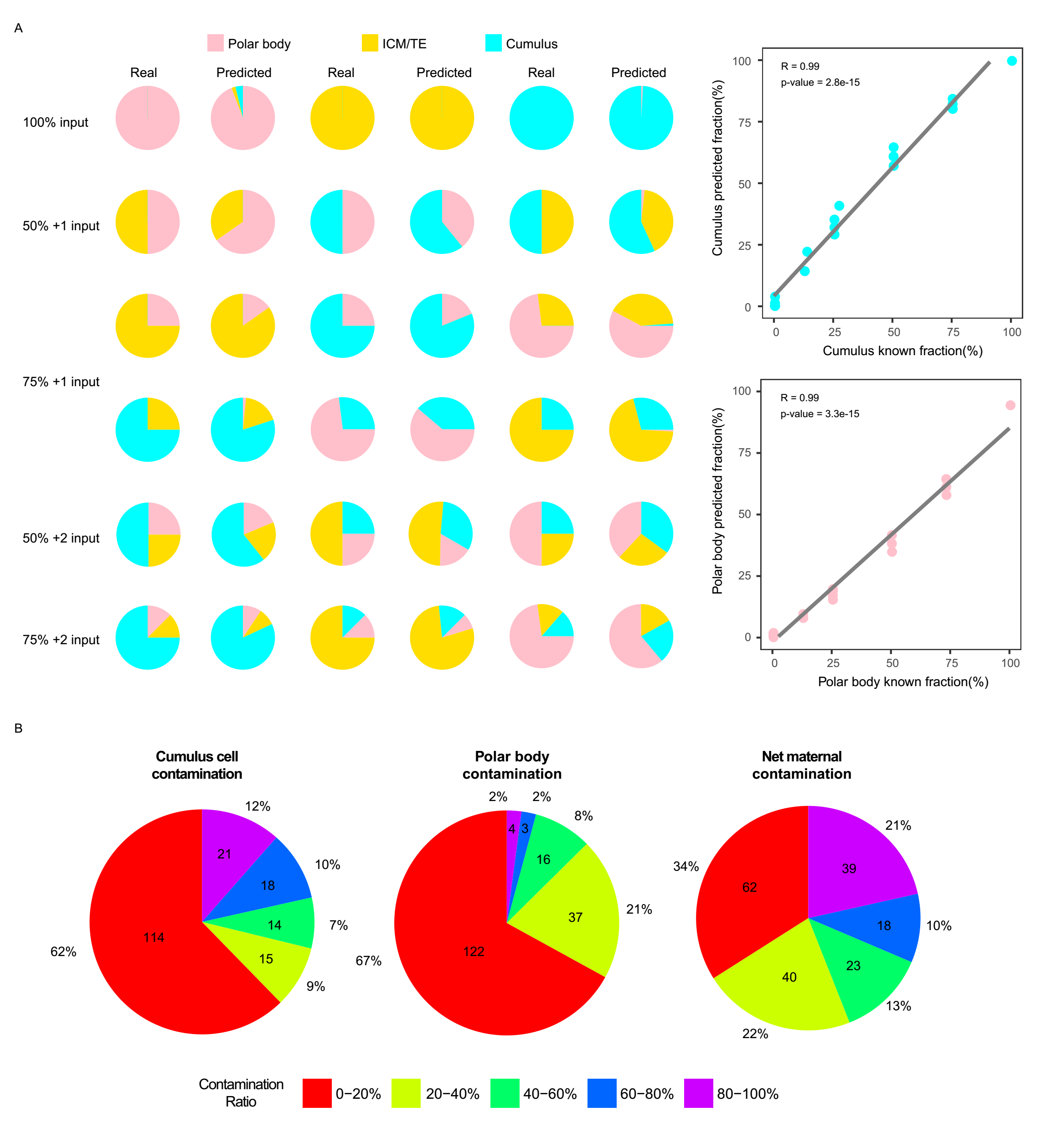
图4 胚胎培养液(SECM)中母源DNA污染率推导。(A)计算机模拟数据验证计算方法的准确性;(B)实际SECM中母源DNA污染比例统计。
4.胚胎培养液游离DNA母源污染与染色体非整倍性的整合分析
DNA甲基化测序可以推断染色体拷贝数(CNV)的情况。该研究在细胞系中评估了单细胞BS-seq推断拷贝数结果的准确性,发现单细胞BS-seq得到的拷贝数和用MALBAC得到的结果一致,同时所需的起始量很低(图 5A)。该研究还探究了胚胎培养液游离DNA母源污染对推断胚胎细胞染色体拷贝数的影响。结果显示染色体拷贝数的性别不一致率和假阴性率均随颗粒细胞、极体细胞和母源净污染率的增加而增加。当净母源污染比例高于60%时,性别不一致率和假阴性率分别增加到100%和75%。因此,当母源严重污染时,由SECM推断的胚胎细胞的CNV就变得不准确。
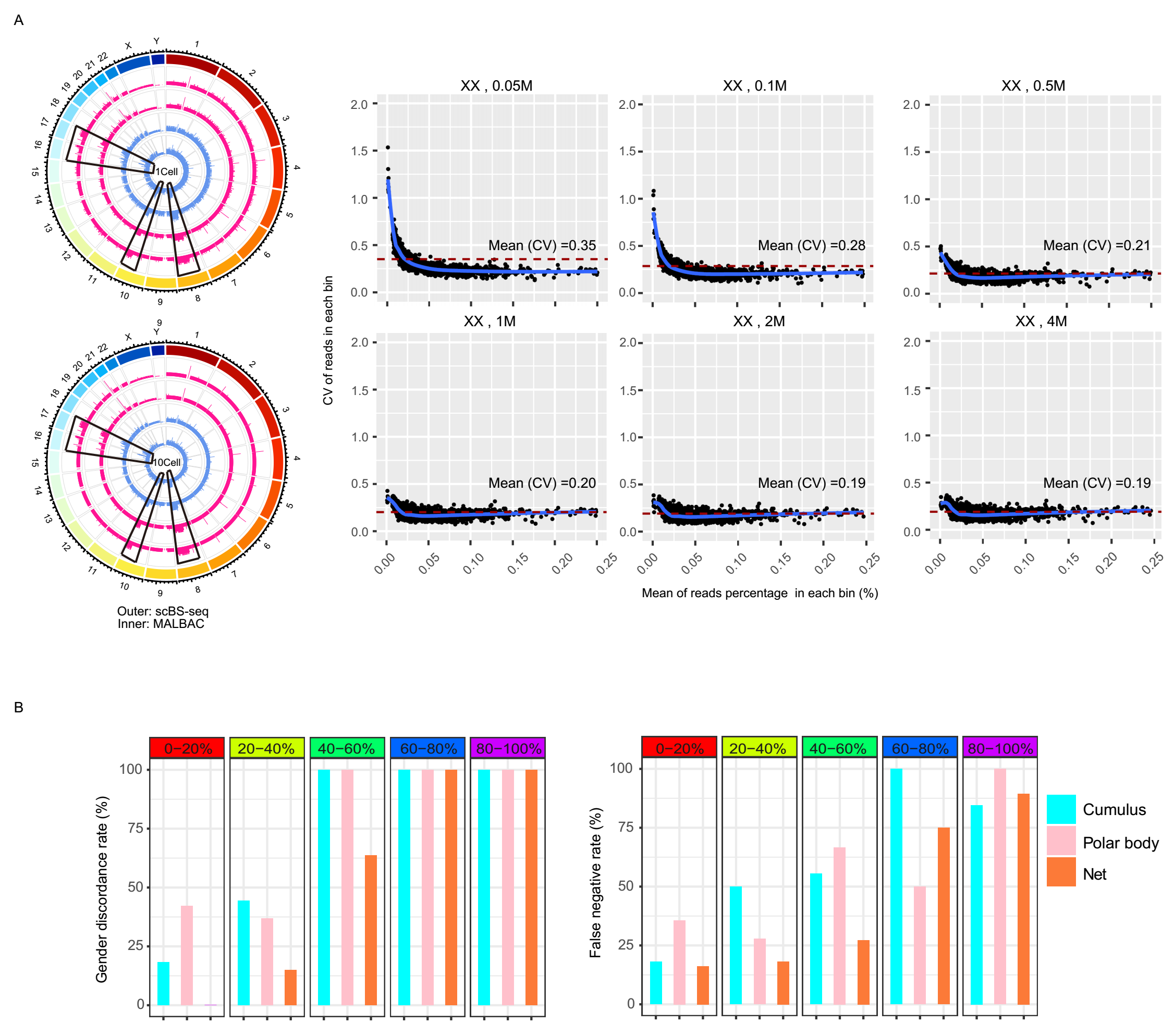
图5 胚胎培养液(SECM)中母源DNA污染和染色体拷贝数的整合分析。(A)scBS-seq推断CNV;(B)母源污染对SECM推断CNV的影响。
综上所述,该研究首次将胚胎培养液中游离DNA的细胞来源追溯到囊胚细胞、颗粒细胞和极体细胞,并且开发了快速便捷定量胚胎培养液游离DNA中母源污染量的算法,同时通过母源污染和染色体非整倍性的整合分析,提高了利用胚胎培养液检测非整倍体胚胎的检测效率。该研究对表征胚胎培养液中游离DNA提供了新见解,为无创植入前胚胎非整倍体遗传学检测(PGT-A)提供了新的视角。
北京大学前沿交叉学科研究院博士研究生陈依东和高原为该研究论文的共同第一作者。北京未来基因诊断高精尖创新中心(ICG)、北京大学生物医学前沿创新中心(BIOPIC)、生命科学学院汤富酬教授、文路副研究员与北京大学第三医院乔杰教授、黄锦副主任医师为该论文的共同通讯作者。该研究项目得到了国家自然科学基金委、生命科学联合中心(CLS)和北京未来基因诊断高精尖创新中心的支持。
论文链接:
https://www.jci.org/articles/view/146051

